3. November 2024Peter Pionke
Neuer, diagnostischer Marker und das Rätsel in der Biologie
 Der Wuppertaler Molekularbiologe- und Zellforscher Prof. Dr. Martin Simon – © Sebastian Jarych
Der Wuppertaler Molekularbiologe- und Zellforscher Prof. Dr. Martin Simon – © Sebastian JarychDer Wuppertaler Molekularbiologe- und Zellforscher Martin Simon hat die diesjährige Nobelpreisvergabe in den Bereichen Medizin und Chemie gespannt mitverfolgt und erklärt die vielfältigen Möglichkeiten, die sich daraus für die Forschung und zukünftige Therapieansätze herleiten. Darüber hat Autor Uwe Blass mit ihm im Rahmen der beliebten Uni-Reihe „Transfergeschichten“ gesprochen.
MicroRNAs
Der Medizinnobelpreis geht 2024 an die US-Amerikaner Victor Ambros und Gary Ruvkun für die Entdeckung der microRNAs (kleine, nicht-codierende RNA Moleküle) und ihre Rolle bei der Genregulierung. „Da fang ich einmal bei den Genen an“, beginnt Simon, „die sind nämlich Teil unseres Erbguts und werden an- und ausgeschaltet. Dadurch kommen die unterschiedlichen Zelltypen in unserem Körper zustande.“
 © Bergische Universität
© Bergische UniversitätSo treten Leberzellen anders in Erscheinung, haben also z. B. ein anderes Genexpressionsmuster als Nieren- Haut-, Epithel- oder Blutzellen. „Wir haben ja 23.000 Gene als Mensch, und es sind immer andere Gene an- und ausgeschaltet, wodurch wir unterschiedliche Organe und Gewebe bilden können. Es muss nun aber irgendwie dem Gen gesagt werden, ob es nun eingeschaltet oder ausgeschaltet sein soll, und das heißt dann, dass aus der DNA ein Protein (Eiweiß) entsteht, oder eben nicht. Dieses An- und Ausschalten nennt man die Genregulation.“
Dabei sei die microRNA ein wesentlicher Bestandteil. Zwar kannte man solche kleine RNA-Stückchen, wie Simon sie nennt, schon lange, jedoch wusste man nichts von ihren genregulatorischen Fähigkeiten und hatte sie unter dem Aspekt ´Abfallprodukt` einsortiert. Als man die microRNA jedoch genauer untersuchte, stellte man irgendwann fest, dass sie die Genregulation negativ beeinflusst. „Ist zu einem Gen eine passende microRNA existent, wird dieses Gen abgeschaltet, es kommt kein Protein da raus. Sie blockiert die Proteinsynthese.“
Um die Bedeutung dieser Entdeckung besser einzuordnen, erklärt Simon, es sei bildlich so, als ob man vor einem Gerät stehe und nicht wisse, wie es funktioniere. Nun habe man aber den ‚Schalter‘ gefunden und könne erklären, warum Dinge so oder so funktionierten.
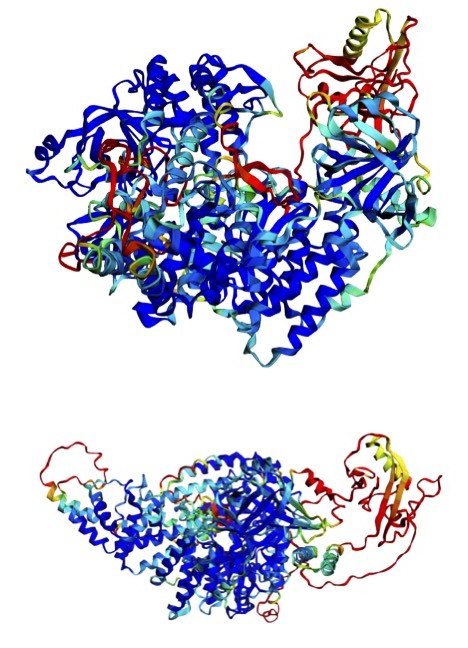 Zwei Perspektiven der 3D-Ansicht der AlphaFold2 berechneten Proteinstruktur einer RNA-abhängigen RNA Polymerase (P. tetraurelia RDR2).
Zwei Perspektiven der 3D-Ansicht der AlphaFold2 berechneten Proteinstruktur einer RNA-abhängigen RNA Polymerase (P. tetraurelia RDR2).„Die Beschreibung der microRNAs hat gezeigt, wie all unsere Zellen das machen und es gibt Schätzungen, die im optimistischsten Fall davon ausgehen, dass ungefähr 60 Prozent unsere Gene darüber reguliert werden. Jetzt können Forscher erklären, warum zum Beispiel dieses oder jenes Protein in der Leberzelle auftaucht, in der Nierenzelle aber nicht, weil man schauen kann, ob die micrRNA die Entstehung dieses Proteins verhindert. Das ist der Schalter, den die beiden Nobelpreisträger gefunden und definiert haben.“
Forschungen am Fadenwurm liefern Erkenntnisse für menschlichen Organismus
Die beiden ausgezeichneten Wissenschaftler fanden microRNA erstmals bei ihrer Forschung am Fadenwurm (C. elegans), der bei vielen Forschungsprojekten eine wichtige Rolle spielt. „Der Fadenwurm ist ein genetischer Modellorganismus, der seit den 1920er Jahren schon bekannt ist. Er hat ein ungeheures Potential, weil es ein Tier mit vielen Zellen ist. Eine Differenzierung der Zelltypen ist bei ihm möglich, er hat einzelne Organe, aber er ist ausreichend primitiv, um ihn leicht untersuchen und auch in großer und schneller Generationszahl kultivieren zu können.“ Ein Würmchen habe gerade mal 800 Zellen und es gebe auch Methoden, jede einzelne Zelle eines Wurmes zu trennen und separat zu analysieren.
„Da hat man dann alle Organe dabei und kann die gruppieren. Das wäre schon mit einer Maus vom Aufwand her nicht mehr machbar. Man nutzt sie auch für toxikologische Analysen oder die Untersuchung von DNA-Schäden. Dieser Wurm ist wirklich für die Forschung ein Allrounder und das hat sich auch schon bei früheren Nobelpreisverleihungen gezeigt.“ Bereits 2006 ist ein Preis an zwei Forscher gegangen, die mit diesem Wurm gearbeitet hatten.
„Das ist also heute der zweite Nobelpreis in der Medizin, der an die Erstbeschreibung an kleinen RNAs an die Arbeit mit C. elegans geht. Alle diese Erkenntnisse, die man bei diesem Wurm gewonnen hat, und das ist das Besondere, kann man auf die medizinischen Aspekte vom Menschen übertragen, und das ist ein besonders gutes Beispiel für die Notwendigkeit von Grundlagenforschung“
 Medizinische Forschung verlängert Leben – © Pixabay
Medizinische Forschung verlängert Leben – © PixabayDurch microRNA kann man Krankheiten besser verstehen
MicroRNAs sind nicht zu verwechseln mit der durch die Covid-Impfstoffe berühmt gewordenen mRNA – der Messenger-RNA, auch als Boten-RNA bekannt. „Durch microRNA kann man Krankheiten nun besser verstehen, weil bei vielen Krankheitsbildern die Genexpression, vor allem beim Krebs, außer Kontrolle geraten ist. Wir beginnen zu verstehen, wie der Krebs sich überhaupt entwickelt und wie es zu der Fehlsteuerung dieser Genexpression kommt, d.h. warum sind Gene, die in der normalen Leberzelle nicht angeschaltet sind, in einem Leberkarzinom nun angeschaltet?“
Mit dieser Erkenntnis könne man dann verstehen, wie sich dieser Krebs entwickelt bzw. warum er so ist, wie er ist. „Wir sehen, dass im Krebs viele microRNAs anders reguliert sind. Es gibt krebsspezifische microRNAs, die dann mehr da sind oder auch weniger da sind. Wichtig ist, dass diese microRNAs hier fehlreguliert sind, denn die Natur hat sie ja nicht gemacht, um Krebs entstehen zu lassen.“
Die Wissenschaftler könnten nun aus diesen Fehlregulationen ableiten, wie schnell oder welche eine spezifische Therapie angegangen werden müsse. „Das ist sehr wichtig für die Therapie, denn vielleicht habe ich noch einen längeren Zeitfaktor, um die Erkrankung lokal zu behandeln oder muss ganz schnell etwas machen, um eine Metastasierung zu verhindern. Somit ist es auch ein diagnostischer Marker geworden.“ Zudem eröffneten die Forschungen auch die Möglichkeit, prognostische Marker zu benutzen, also im Vorfeld zu handeln.
„Ein Beispiel für so einen prognostischen Marker wäre die microRNA106b, denn eine genetisch bedingte Veränderung in dieser RNA geht mit einem höheren Risiko für ein Leberkarzinom einher. Wir haben in der microRNA einen prognostischen Marker, einen diagnostischen Marker und wir haben einen Ansatz, an diesen Stellen einzugreifen und somit ein therapeutisches Werkzeug gewonnen. Wir können microRNAs blocken oder zu Zellen hinzugeben, welche diese nicht mehr herstellen. Man kann von außen in dieses RNA-System eingreifen und die regulatorische Funktion beeinflussen, also „wir können den Schalter selbst betätigen“. Und seit 2017 gibt es in der EU bereits neun Zulassungen für Wirkstoffe auf Basis von solcher regulatorischer RNA.“
 Die begehrte Nobelpreis-Medaille – © gemeinfrei
Die begehrte Nobelpreis-Medaille – © gemeinfreiEine neue Welt der Proteinstrukturen
In der gleichen Woche, in der der Medizinnobelpreis verliehen wurde, verkündete das Komitee auch den Preis für Chemie an die Wissenschaftler David Baker (USA) sowie Demis Hassabis und John Jumper (Großbritannien). Baker hat bereits 2003 ein Computerprogramm entwickelt, welches neue Proteine entwerfen konnte und damit eine neue Welt von Proteinstrukturen eröffnet.
„Proteine an sich sind ja der wesentliche Bestandteil des Lebens, d.h. sie sind eine der Hauptkomponenten der Zellen. Sie bieten eine Gerüststruktur und sie bieten enzymatische Aktivität, um Substanzen im Stoffwechsel umzusetzen. Alle Proteine sind in unterschiedlichen Kombinationen aus 20 Bausteinen aufgebaut, das sind die Aminosäuren, und die werden in einer linearen Kette aneinandergehängt. Jetzt gibt es Aminosäuren, die sind groß, andere sind klein, manche sind positiv geladen, andere sind negativ geladen.“
Logischerweise sei die Zusammensetzung dieser Aminosäuren ausschlaggebend für die Eigenschaften des gesamten Proteins. Wenn man nun aber eine positive und eine negative Aminosäure habe, bleibe das Protein nicht linear, sondern falte sich zusammen. Dadurch entstehe eine dreidimensionale Struktur, die in der Biologie lange ein Rätsel war.
„Wenn man sich vorstellt, dass es Proteine gibt, die aus mehreren tausend Aminosäuren aufgebaut sind, könnte es rein theoretisch so viele verschiedene Wechselwirkungen zwischen diesen einzelnen Bausteinen geben. Aber man muss die Struktur kennen um die Funktion des Proteins zu verstehen. Das war früher ein riesiger Aufwand. Man musste die Proteine kristallisieren, also eindampfen und eine Kristallstruktur entwickeln. Und dann konnte man über eine Röntgenstrukturanalyse oder eine NMR-Analyse erfahren, wie dieses einzelne Protein gefaltet ist, d.h. man kann ein 3D-Modell erstellen.“
 Weltweit wird nach wirksamen Wirkstoffen gegen Krebs geforscht – © Pixabay
Weltweit wird nach wirksamen Wirkstoffen gegen Krebs geforscht – © PixabaySelbst bei diesem aufwändigen Verfahren ließen sich auch nach mehreren Jahren Arbeit nicht immer alle Proteine kristallisieren. Interessant in diesem Zusammenhang ist die Tatsache, dass das Nobelpreiskommitee bereits in der Vergangenheit mehrmals Preise zu Forschungsarbeiten um die chemische Analyse der Proteinstrukturen verliehen habe. Mit der diesjährigen Verleihung komme aber nun ein ganz anderer Ansatz zum Tragen. „Man benutzt ein KI-basiertes Tool, um diese Wechselwirkungen zu berechnen“, erklärt Simon.
Der Wissenschaftler weiter: „Das Tool heißt ‚AlphaFold‘, ist eine KI-basierte Software, die die Struktur von Proteinen basierend auf der Aminosäuresequenz, also auf der linearen Sequenz, voraussagt. Dieses Tool ist auch selbstkorrigierend, kontrolliert also selber noch einmal das Ergebnis und verbessert seine Voraussage nach und nach immer weiter.“ Diese Software mit bereits bekannten Strukturen trainiert und kontrolliert. Das phantastische an dieser Software sei vor allem, dass sie Open Source veröffentlicht wurde, d.h. sie ist kostenfrei sowohl für die Forschung als auch für die Wirtschaft nutzbar.
„Auch wir können schon damit arbeiten. Wir können unsere Aminosäuresequenzen eingeben und bekommen nach ein wenig Rechenzeit die Struktur heraus. Jetzt gibt es auch schon Folgeprogramme. ´AlphaFold 3` kann z.B. auch berechnen, ob zwei verschiedene Proteine miteinander agieren und aneinanderbinden, man kann also schon in die nächste Dimension gehen. Das ist ein riesiger Fortschritt. Wir haben es installiert und benutzen es mittlerweile permanent.“
Das Programm funktioniert wie folgt: der Nutzer gibt eine Buchstabenfolge seiner Aminosäuresequenzen ein und der Computer zeigt nach einer Berechnungszeit die dreidimensionale bildliche Struktur, die man dann auch in alle Richtungen drehen kann. „Früher wäre das die Arbeit einer ganzen Promotion gewesen“, lacht der Forscher. Dadurch werde die Forschung um ein Vielfaches beschleunigt und inzwischen sei es auch so, dass man in den Genomdatenbanken diese Strukturen schon für alle Gene schon mit hinterlegt habe.
KI zeigt auch, wo sie unsicher ist
Die Wissenschaftler Hassabis und Jumper haben eine gigantische Datenbank erstellt. Diese Datenbank könnte u.a. auch bei der Klärung von Antibiotika-Resistenzen helfen oder beim Einsatz von Enzymen zum Abbau von Kunststoffen.
 Ein medizinisches Forschungslabor – © Pixabay
Ein medizinisches Forschungslabor – © Pixabay„Man kann also nun in einer Art Designarbeit Stoffe mit der Software bauen lassen“, sagt der Fachmann. Zwar versteht wie so oft der Anwender nicht komplett, was die KI tue. „Bei ‚AlphaFold‘ ist es da aber z.B. so, dass die Bereiche, in denen sich der Algorithmus nicht sicher ist, eingefärbt werden. Blau meint, da ist er sich sicher und rot meint, da ist er sich nicht so sicher. Das ist ein wesentlicher Faktor, den man auch als Qualitätskriterium nennen muss. Das Programm hinterlässt im Unsicherheitsfalle damit eine Markierung.“
Wissenschaft schafft Impulse für die Zukunft
Nobelpreise bedeuten immer eine Würdigung der Wissenschaftler*innen und deren Forschung. Was aber bedeuten diese Entdeckungen für tägliche Arbeit?
„Wir reden ja von einem Transfer von Grundlagenforschung, d.h. es hat bereits zwei Mal den Medizinnobelpreis gegeben für C. elegans-Forscher, deren Ergebnisse auf den Menschen übertragbar sind.“ Die Nobelpreiswürdigung schaffe mit diesen Auslobungen neue Impulse für die Wissenschaft. „Mit dieser Würdigung wird ein Transfer zu neuen Aspekten in die Medizin hineingetragen“, sagt Simon abschließend.
Er betont: „Wir brauchen neue Ansatzideen. Genauso wie bei den Antibiotika. Da haben wir zwar eine Weiterentwicklung der bestehenden Antibiotika, die immer weiter modifiziert werden, um den Resistenzen auszuweichen, aber wir sind mit den klassischen Antibiotika in einer fast toten Zone. Wir können nur dran herummodifizieren, aber wir rennen immer wieder in die nächste Antibiotikaresistenz hinein. Dann braucht man in diesen Feldern wieder einen neuen Impuls. Und das können aus der Grundlagenforschung z.B. Viren sein, die Bakterien infizieren. Wenn wir die dann modifizieren, kann man wieder neue Wege gehen. Beim Chemienobelpreis wiederum ist es der Aspekt, dass diese neue Software ‚AlphaFold‘ unser tägliches Leben in der Grundlagenforschung tatsächlich bereits verändert hat, weil wir schneller und kostengünstiger an solide Ergebnisse kommen.“
Uwe Blass
 Prof. Dr. Martin Simon – © Sebastian Jarych
Prof. Dr. Martin Simon – © Sebastian Jarych
Über Prof. Dr. Martin Simon
Prof. Dr. Martin Simon leitet die Fachgruppe Molekulare Zellbiologie und Mikrobiologie in der Fakultät Mathematik und Naturwissenschaften an der Bergischen Universität.
Weiter mit:




Kommentare
Neuen Kommentar verfassen